|
0 M! N$ o" g) p# ^& J4 g' K жө·жҙӢеҹәеӣ з»„еӯҰзҷҪзҡ®д№ҰпјҲ2021/11/03пјү 3 P) E1 x! G0 L
///////////////////// 3 C6 S$ @' V8 P5 a2 S, H4 y' Y1 ^
иҺ·еҸ–е…Ёзҗғз”ҹзү©йҒ—дј дҝЎжҒҜеҸҜд»ҘеҠ ж·ұжҲ‘们еҜ№з”ҹе‘Ҫзҡ„зҗҶи§ЈгҖӮ2017е№ҙпјҢе…Ёзҗғ科еӯҰ家еҗҲдҪңеҗҜеҠЁдәҶеӨ§и§„жЁЎең°зҗғз”ҹзү©еҹәеӣ з»„и®ЎеҲ’пјҲEBPпјүпјҢиҜҘйЎ№зӣ®ж—ЁеңЁеҚҒе№ҙеҶ…еҜ№еӨ§зәҰ150 дёҮз§Қе·ІзҹҘзңҹж ёз”ҹзү©иҝӣиЎҢжөӢеәҸгҖӮ2018е№ҙпјҢPNASжқӮеҝ—еҸ‘иЎЁж–Үз« д»Ӣз»ҚдәҶиҜҘи®ЎеҲ’зҡ„иҜҰз»ҶеҶ…е®№гҖӮиҜҘи®ЎеҲ’еҢ…еҗ«еӨҡдёӘеӯҗйЎ№зӣ®пјҢеӨ§йғЁеҲҶеӯҗйЎ№зӣ®йғҪеңЁйЎәеҲ©иҝӣиЎҢдёӯгҖӮдҫӢеҰӮпјҢ10KPйЎ№зӣ®е·Із»ҸиҺ·еҫ—дәҶеӨ§зәҰ1,500дёӘзү©з§Қзҡ„еҹәеӣ з»„еәҸеҲ—гҖӮ2020е№ҙпјҢB10KйЎ№зӣ®еңЁNatureдёҠеҸ‘иЎЁдәҶ360еӨҡеҸӘйёҹзұ»зү©з§Қеҹәеӣ з»„жөӢеәҸеҲҶжһҗзҡ„жңҖж–°з ”з©¶жҲҗжһңгҖӮ
# N2 y+ O' U5 h. Q; c& q+ {% d4 A$ T z8 c0 [& t
еңЁжө·жҙӢз”ҹзү©еҹәеӣ з»„еӯҰж–№йқўпјҢйқ’еІӣеҚҺеӨ§еҹәеӣ з ”з©¶йҷўеңЁжң¬ж¬ЎICG-16жңҹй—ҙпјҢйӣҶдёӯеҸ‘еёғдәҶ4дёӘжө·жҙӢз”ҹзү©еҹәеӣ з»„ж•°жҚ®еә“пјҢеҲҶеҲ«дёәж°ҙз”ҹи„ҠжӨҺеҠЁзү©еҹәеӣ з»„гҖҒи—»зұ»еҹәеӣ з»„гҖҒжө·жҙӢж— и„ҠжӨҺеҠЁзү©д»ҘеҸҠжө·жҙӢеҫ®з”ҹзү©еҹәеӣ з»„гҖӮ
+ z d* q- }+ z/ c* l ///////////////////// 7 C9 b8 b3 u3 @% o6 `5 N+ y2 l
йқ’еІӣеҚҺеӨ§еҹәеӣ з ”з©¶йҷўжһ„е»әдәҶеӣӣдёӘжө·жҙӢз”ҹзү©еҹәеӣ з»„еӯҰж•°жҚ®еә“пјҢ并еҹәдәҺиҝҷдәӣж•°жҚ®еә“иҝӣиЎҢдәҶеҹәеӣ з»„еӯҰеҲҶжһҗпјҢзӣёе…із»“жһңе·ІеҲҶеҲ«еңЁbioRixvдёҠйў„еҚ°гҖӮ ! O0 j# a2 @% S* t
01 ж°ҙз”ҹи„ҠжӨҺеҠЁзү©еҹәеӣ з»„ж•°жҚ®еә“
6 E7 t, _8 m; ^; [' ?' E / ?3 z/ [# K0 A. m; w8 F1 F
ж°ҙз”ҹи„ҠжӨҺеҠЁзү©дё»иҰҒзҡ„з ”з©¶еҜ№иұЎйҖҡеёёеҢ…жӢ¬йұјзұ»гҖҒдёӨж –еҠЁзү©гҖҒзҲ¬иЎҢеҠЁзү©е’Ңжө·жҙӢе“әд№іеҠЁзү©пјҢеңЁз”ҹжҖҒгҖҒз»ҸжөҺе’Ң科еӯҰдёҠеҜ№е…·жңүеҫҲй«ҳзҡ„з§‘з ”д»·еҖјгҖӮзӣ®еүҚж°ҙз”ҹи„ҠжӨҺз”ҹзү©е·Із»ҸжңүеӨҡдёӘзү©з§Қиў«жөӢеәҸпјҢдҪҶжҳҜзӣёжҜ”йҷҶз”ҹи„ҠжӨҺеҠЁзү©пјҢе…¶иў«йҮҚи§ҶзЁӢеәҰдҫқ然дёҚи¶ігҖӮеңЁеҹәеӣ з»„ж•°жҚ®иө„жәҗж–№йқўд№ҹиҝҳжІЎжңүзӣёе…іж•°жҚ®еә“еҜ№е·Із»ҸжөӢеәҸзү©з§ҚиҝӣиЎҢе…ЁйқўжҖ»з»“гҖӮеӣ жӯӨпјҢйқ’еІӣеҚҺеӨ§еҹәеӣ з ”з©¶йҷўеңЁеҹәдәҺFish10KйЎ№зӣ®еҲқжӯҘз»“жһңеҹәзЎҖдёҠпјҢжұҮжҖ»ж¶өзӣ–иҪҜйӘЁйұјгҖҒзЎ¬йӘЁйұјгҖҒдёӨж –гҖҒзҲ¬иЎҢгҖҒжө·жҙӢе“әд№ізӯү5еӨ§зұ»ж°ҙз”ҹи„ҠжӨҺеҠЁзү©пјҢе…ұ630з§Қзү©з§Қзҡ„еҹәеӣ з»„дҝЎжҒҜпјҲеӣҫ1пјүпјҢ并иҝӣиЎҢжҜ”иҫғеҹәеӣ з»„еӯҰеҲҶжһҗпјҢд»ҘжҺўеҜ»ж°ҙз”ҹи„ҠжӨҺеҠЁзү©еңЁзү©з§Қжј”еҢ–е’ҢзҺҜеўғйҖӮеә”жҖ§ж–№йқўзҡ„еҲҶеӯҗжңәеҲ¶гҖӮ
' w/ i" J a5 q- ^& Y6 P+ V, j еӣҫ1. 630з§Қж°ҙз”ҹи„ҠжӨҺеҠЁзү©еҹәеӣ з»„иө„жәҗзҡ„дё»иҰҒеҲҶж”ҜйҖҡиҝҮеҜ№еҹәеӣ з»„еӨ§е°Ҹе’ҢGCеҗ«йҮҸиҝӣиЎҢеҲҶжһҗпјҢеҸ‘зҺ°дёӨж –еҠЁзү©еңЁеҹәеӣ з»„еӨ§е°ҸдёҠжңүжҳҫи‘—зҡ„е·®ејӮпјҲеӣҫ2пјҢеӣҫ3пјүгҖӮдёӨж –зұ»зү©з§Қз»„иЈ…зҡ„еҹәеӣ з»„иҢғеӣҙдёә0.5 Gb ~ 32 GbгҖӮеҗҢж ·еңЁйұјзұ»дёӯд№ҹи§ӮеҜҹеҲ°еҹәеӣ з»„еӨ§е°ҸеҸҳеҢ–пјҢе…¶еҹәеӣ з»„еӨ§е°ҸиҢғеӣҙеҲҶеёғеңЁ350 Mb ~ 40 GbгҖӮеңЁиҪҜйӘЁйұјдёӯпјҢеҹәеӣ з»„жҷ®йҒҚи¶…иҝҮ3GпјҢе…¶еӨ§е°ҸиҢғеӣҙдёә3 Gb ~ 6.7 GbгҖӮ
% h' P9 a2 C, v1 f! B еӣҫ2. ж°ҙз”ҹи„ҠжӨҺеҠЁзү©еҹәеӣ з»„зҡ„з»„иЈ…иҙЁйҮҸе’ҢеӨ§е°Ҹеӣҫ2. ж°ҙз”ҹи„ҠжӨҺеҠЁзү©еҹәеӣ з»„зҡ„з»„иЈ…иҙЁйҮҸе’ҢеӨ§е°Ҹ ; R/ I5 b* u. ~8 X2 e# x
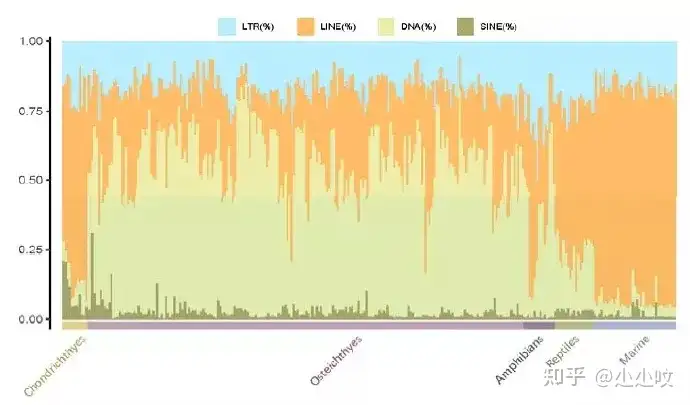
еӣҫ3. GCеҗ«йҮҸе’ҢTEsеңЁж°ҙз”ҹи„ҠжӨҺеҠЁзү©еҹәеӣ з»„дёӯзҡ„жҜ”дҫӢеҲҶеёғжҜ”иҫғеҹәеӣ з»„еӯҰеҲҶжһҗиЎЁжҳҺпјҢиҪ¬еә§еӯҗе…ғ件дёӯLINEзұ»еһӢеңЁиҪҜйӘЁйұјдёӯеҚ дјҳеҠҝпјҢDNAзұ»еһӢиҪ¬еә§еӯҗиҫғе°‘пјӣеңЁзЎ¬йӘЁйұјеҪ“дёӯпјҢLINEе’ҢDNAиҪ¬еә§еӯҗзҡ„жҜ”дҫӢеҸ‘з”ҹдәҶж”№еҸҳпјҢеҚіDNAиҪ¬еә§еӯҗжҲҗдёәдјҳеҠҝзұ»еһӢпјӣйҡҸзқҖзү©з§Қжј”еҢ–пјҢеңЁдёӨж –е’Ңе“әд№іеҠЁзү©дёӯпјҢLINEеҸҲжҲҗдёәдё»иҰҒйҮҚеӨҚеәҸеҲ—зұ»еһӢпјҲеӣҫ4пјүгҖӮTEеңЁз”ҹзү©жј”еҢ–дёӯзҡ„з ”з©¶д№ӢеүҚдёҖзӣҙе°‘жңүжҠҘйҒ“пјҢиҝҷж¬ЎйҖҡиҝҮеҜ№ж°ҙз”ҹи„ҠжӨҺеҠЁзү©еҹәеӣ з»„иҝӣиЎҢеӨ§ж•°жҚ®еҲҶжһҗиЎЁжҳҺTEеҗ«йҮҸзҡ„еҸҳеҢ–дҝғиҝӣдәҶз”ҹзү©д»Һж°ҙз”ҹеҲ°йҷҶз”ҹзҡ„жј”еҢ–е’ҢеҜ№зҺҜеўғзҡ„йҖӮеә”жҖ§гҖӮ 8 P5 J4 x1 T9 Z8 Q- \
 еҗҢж—¶йҖҡиҝҮиҜҘж•°жҚ®еә“пјҢеҜ№и„ҠжӨҺеҠЁзү©ж°ҙз”ҹеҲ°йҷҶз”ҹиҪ¬жҚўе…ій”®еҹәеӣ 家ж—Ҹд№ҹиҝӣиЎҢдәҶеҲҶжһҗпјҲеӣҫ5пјүгҖӮиҝҷд№ҹж”№еҸҳдәҶд»ҘеҫҖеҜ№еҹәеӣ еҠҹиғҪзҡ„з ”з©¶еҸӘиғҪеҹәдәҺе°‘ж•°зү©з§Қж•°жҚ®зҡ„еұҖйқўгҖӮеңЁи„ҠжӨҺеҠЁзү©д»Һж°ҙеҲ°йҷҶзҡ„иҝӣеҢ–иҝҮзЁӢдёӯпјҢйҮҚиҰҒзҡ„еҸҳеҢ–д№ӢдёҖжҳҜе‘јеҗёж–№ејҸпјҢеӣ жӯӨжҲ‘д»¬з ”з©¶дәҶе—…и§үеҸ—дҪ“ORеҹәеӣ 家ж—ҸгҖӮз»“жһңиЎЁжҳҺж°ҙжә¶жҖ§зҡ„ORsеҹәеӣ еӨ§йҮҸеҮәзҺ°еңЁзЎ¬йӘЁйұјдёӯпјҢиҖҢж°”жә¶жҖ§зҡ„ORsеңЁйҷҶз”ҹи„ҠжӨҺеҠЁзү©пјҲдёӨж –гҖҒзҲ¬иЎҢгҖҒйҷҶз”ҹе“әд№ізұ»пјүдёӯеҲҷеӨ§йҮҸеҮәзҺ°пјӣжӣҙжңүи¶Јзҡ„жҳҜпјҢиҪҜйӘЁйұјдёӯзјәе°‘жүҖжңүType1е’ҢйғЁеҲҶType2 зҡ„ORsпјҢиҝҷйңҖиҰҒиҝӣдёҖжӯҘйӘҢиҜҒзӣёе…іеҹәеӣ зҡ„еҠҹиғҪгҖӮеҗҢж—¶йҖҡиҝҮиҜҘж•°жҚ®еә“пјҢеҜ№и„ҠжӨҺеҠЁзү©ж°ҙз”ҹеҲ°йҷҶз”ҹиҪ¬жҚўе…ій”®еҹәеӣ 家ж—Ҹд№ҹиҝӣиЎҢдәҶеҲҶжһҗпјҲеӣҫ5пјүгҖӮиҝҷд№ҹж”№еҸҳдәҶд»ҘеҫҖеҜ№еҹәеӣ еҠҹиғҪзҡ„з ”з©¶еҸӘиғҪеҹәдәҺе°‘ж•°зү©з§Қж•°жҚ®зҡ„еұҖйқўгҖӮеңЁи„ҠжӨҺеҠЁзү©д»Һж°ҙеҲ°йҷҶзҡ„иҝӣеҢ–иҝҮзЁӢдёӯпјҢйҮҚиҰҒзҡ„еҸҳеҢ–д№ӢдёҖжҳҜе‘јеҗёж–№ејҸпјҢеӣ жӯӨжҲ‘д»¬з ”з©¶дәҶе—…и§үеҸ—дҪ“ORеҹәеӣ 家ж—ҸгҖӮз»“жһңиЎЁжҳҺж°ҙжә¶жҖ§зҡ„ORsеҹәеӣ еӨ§йҮҸеҮәзҺ°еңЁзЎ¬йӘЁйұјдёӯпјҢиҖҢж°”жә¶жҖ§зҡ„ORsеңЁйҷҶз”ҹи„ҠжӨҺеҠЁзү©пјҲдёӨж –гҖҒзҲ¬иЎҢгҖҒйҷҶз”ҹе“әд№ізұ»пјүдёӯеҲҷеӨ§йҮҸеҮәзҺ°пјӣжӣҙжңүи¶Јзҡ„жҳҜпјҢиҪҜйӘЁйұјдёӯзјәе°‘жүҖжңүType1е’ҢйғЁеҲҶType2 зҡ„ORsпјҢиҝҷйңҖиҰҒиҝӣдёҖжӯҘйӘҢиҜҒзӣёе…іеҹәеӣ зҡ„еҠҹиғҪгҖӮ # ?) Z5 x" D7 t9 t; c% p
еӣҫ5. 62дёӘд»ЈиЎЁзү©з§Қзҡ„е—…и§үеҸ—дҪ“зҡ„еҹәеӣ ж•°пјҢдёӨж –зұ»гҖҒзҲ¬иЎҢзұ»е’Ңжө·жҙӢе“әд№іеҠЁзү©еӣҫ5. 62дёӘд»ЈиЎЁзү©з§Қзҡ„е—…и§үеҸ—дҪ“зҡ„еҹәеӣ ж•°пјҢдёӨж –зұ»гҖҒзҲ¬иЎҢзұ»е’Ңжө·жҙӢе“әд№іеҠЁзү©в–¶ йў„еҚ°ж–Үз« дҝЎжҒҜпјҡ % r! b: _" F" U2 d
https://biorxiv.org/cgi/content/short/2021.10.29.466026v1 ! o$ z/ g( l' S8 W( }: {
в–¶ зӣёе…іж•°жҚ®йӣҶдҝЎжҒҜпјҡ " U {+ |! c. r. F4 h* ^: O7 E
3 y7 t( J" S6 p* V0 I1 L! o 02 и—»зұ»еҹәеӣ з»„ж•°жҚ®еә“
, X; G5 n9 Q$ m6 Y W * M: t) f6 ]! w$ ?
и—»зұ»е…·жңүй«ҳеәҰеӨҡж ·зҡ„еҲҶзұ»еӯҰе’ҢеҪўжҖҒеӯҰзү№еҫҒпјҢе№ҝжіӣз”ЁдәҺз ”з©¶жӨҚзү©иө·жәҗе’ҢйҷҶең°еҢ–д»ҘеҸҠеӨҡз»ҶиғһиҝӣеҢ–гҖӮи—»зұ»еҹәеӣ з»„еӯҰз ”з©¶зҡ„жңҖеӨ§жҢ‘жҲҳд№ӢдёҖжҳҜеҫ®з”ҹзү©зҡ„жұЎжҹ“пјҢе°Өе…¶жҳҜе…ұз”ҹеҫ®з”ҹзү©гҖӮжҲ‘д»¬ж— жі•еңЁи—»зұ»ж ·е“ҒеҮҶеӨҮиҝҮзЁӢдёӯе®Ңе…ЁеҺ»йҷӨеҫ®з”ҹзү©пјҢиҝҷз»ҷи—»зұ»еҹәеӣ з»„зҡ„з»„иЈ…еёҰжқҘжһҒеӨ§зҡ„жҢ‘жҲҳгҖӮзӣ®еүҚе…¬ејҖзҡ„и—»зұ»еҹәеӣ з»„жңү190еӨҡз§ҚпјҢе…¶дёӯеӨ§йғЁеҲҶжҳҜз»ҝи—»гҖӮйҖҡиҝҮжҜ”иҫғдёҚеҗҢи—»зұ»д№Ӣй—ҙзҡ„жұЎжҹ“еҫ®з”ҹзү©з»„жҲҗпјҢеҸ‘зҺ°дёҚеҗҢзұ»еһӢи—»зұ»зҡ„е…ұз”ҹеҫ®з”ҹзү©з§Қзұ»зӣёдјјпјҢдҪҶеҜ№дәҺзү№е®ҡзҡ„и—»зұ»еӯҳеңЁдёҖдәӣзү№жңүзұ»еһӢзҡ„еҫ®з”ҹзү©гҖӮеҗҢж—¶зі»з»ҹең°жҜ”иҫғдәҶиҝҷдәӣдёҚеҗҢеҲҶж”Ҝзҡ„и—»зұ»еҹәеӣ з»„зү№еҫҒе·®ејӮпјҢеҢ…жӢ¬еҹәеӣ з»„еӨ§е°ҸгҖҒз»„иЈ…иҝһз»ӯжҖ§е’Ңе®Ңж•ҙжҖ§гҖҒGC еҗ«йҮҸгҖҒйҮҚеӨҚеәҸеҲ—зҡ„дё°еәҰе’ҢиӣӢзҷҪиҙЁзј–з Ғеҹәеӣ ж•°йҮҸпјҲеӣҫ6пјүгҖӮеҹәдәҺ193дёӘи—»зұ»еҹәеӣ з»„и—»зұ»еҹәеӣ з»„ж•°жҚ®пјҢеҲ©з”ЁBUSCOеҚ•жӢ·иҙқеҹәеӣ жһ„е»әдәҶи—»зұ»зі»з»ҹеҸ‘иӮІж ‘пјҲеӣҫ7пјүгҖӮйҮҚе»әзҡ„и—»зұ»зҡ„зі»з»ҹеҸ‘иӮІж ‘дёӯпјҢзҒ°и—»жҳҜжңҖеҸӨиҖҒзҡ„зҡ„и—»зұ»еҲҶж”ҜпјҢеҗҢж—¶з»ҝи—»е’Ңзәўи—»еҸҜд»Ҙжё…жҷ°ең°еҲҶдёәдёӨдёӘдёҚеҗҢзҡ„зҫӨдҪ“пјҢиҖҢиҪ®и—»жҳҜйҷҶз”ҹжӨҚзү©зҡ„иө·жәҗгҖӮ
0 X0 @% ^0 u0 O0 T; N; Q% y% ^ еӣҫ6. е·ІеҸ‘иЎЁзҡ„и—»зұ»еҹәеӣ з»„еҲҶеёғеӣҫ7. и—»зұ»зі»з»ҹеҸ‘иӮІж ‘еӣҫ7. и—»зұ»зі»з»ҹеҸ‘иӮІж ‘в–¶ йў„еҚ°ж–Үз« дҝЎжҒҜпјҡ ( ]2 b: K" e/ S+ g) l
https://biorxiv.org/cgi/content/short/2021.10.30.466624v1
( R$ x8 ?- g7 ]# S. ^6 E; I в–¶ зӣёе…іж•°жҚ®йӣҶдҝЎжҒҜпјҡ 4 W6 `0 b- d* q3 M6 L1 L/ E( I
$ x0 i6 z+ P8 w) ] 03 жө·жҙӢж— и„ҠжӨҺеҠЁзү©еҹәеӣ з»„ж•°жҚ®еә“
" |6 j0 D, a: I+ H. n, u6 `
8 A- P! n, t4 S( K' B еҸ‘еёғзҡ„жө·жҙӢж— и„ҠжӨҺеҠЁзү©еҹәеӣ з»„ж•°жҚ®еә“дёӯпјҢ收йӣҶеҲ°дәҶеӨ§зәҰ 190 дёӘжө·жҙӢж— и„ҠжӨҺеҠЁзү©еҹәеӣ з»„пјҲеӣҫ8пјүгҖӮйҖҡиҝҮеҲ©з”Ё 974 дёӘBUSCOеҚ•жӢ·иҙқеҹәеӣ йҮҚе»әдәҶжө·жҙӢж— и„ҠжӨҺеҠЁзү©зі»з»ҹеҸ‘иӮІж ‘пјҢеҜ№дёҖдәӣй•ҝжңҹеӯҳеңЁдәүи®®зҡ„иҝӣеҢ–е…ізі»иҝӣиЎҢйӘҢиҜҒгҖӮ 4 t3 E; H* h! u6 U, E8 d
еӣҫ8. жө·жҙӢж— и„ҠжӨҺеҠЁзү©еҹәеӣ з»„еҲҶеёғе°Ҫз®Ўж— и„ҠжӨҺеҠЁзү©зҡ„зҘһз»Ҹзі»з»ҹиў«и®ӨдёәжҜ”и„ҠжӨҺеҠЁзү©зҡ„зҘһз»Ҹзі»з»ҹз®ҖеҚ•еҫ—еӨҡпјҢдҪҶдёҚеҗҢй—Ёзұ»зҡ„зҘһз»Ҹзі»з»ҹеӨҚжқӮзЁӢеәҰе·®еҲ«д№ҹиҫғеӨ§гҖӮдҫӢеҰӮпјҢж°ҙжҜҚжӢҘжңүжңҖз®ҖеҚ•зҡ„зҘһз»Ҹзі»з»ҹпјҢз§°дёәвҖңзҘһз»ҸзҪ‘вҖқпјҢжІЎжңүжҳҺжҳҫзҡ„дёӯжһўзҘһз»ҸеҢәеҹҹгҖӮдҪҶжҳҜдёҖдәӣиҪҜдҪ“еҠЁзү©иҝӣеҢ–еҮәдәҶдёӯжһўзҘһз»Ҹзі»з»ҹгҖӮеӣ жӯӨпјҢе®ғ们жҳҜз ”з©¶зҘһз»Ҹзі»з»ҹиҝӣеҢ–зҡ„зҗҶжғізҫӨдҪ“гҖӮжҲ‘们关注йүҙе®ҡдәҶдёүдёӘдёӯжһўзҘһз»ҸеҸ‘иӮІзӣёе…іеҹәеӣ 家ж—ҸгҖӮжӯЈеҰӮйў„жңҹзҡ„йӮЈж ·пјҢеңЁеҹәйғЁж үж°ҙжҜҚдёӯпјҢеҮ д№ҺжІЎжңүеҸ‘зҺ°зӣ®ж Үеҹәеӣ гҖӮжңүи¶Јзҡ„жҳҜпјҢйҷӨдәҶ PCDHa д№ӢеӨ–зҡ„жүҖжңү PCDH еҹәеӣ еңЁз« йұјдёӯйғҪз»ҸеҺҶдәҶжҳҫзқҖзҡ„жү©еўһпјҢиҝҷе°ұжҳҜз« йұјеҰӮжӯӨиҒӘжҳҺзҡ„еҺҹеӣ пјҲеӣҫ9пјүгҖӮ 4 q/ c6 G. C" T2 L9 g
еӣҫ9. ж— и„ҠжӨҺеҠЁзү©зҘһз»Ҹзі»з»ҹеҸ‘иӮІзӣёе…іеҹәеӣ жӢ·иҙқж•°в–¶ йў„еҚ°ж–Үз« дҝЎжҒҜпјҡ 2 y: `% m; C/ X6 H$ ]9 {1 i1 s
https://biorxiv.org/cgi/content/short/2021.10.31.465852v1 4 s2 t, @4 y! d a% P. P- a
в–¶ зӣёе…іж•°жҚ®йӣҶдҝЎжҒҜпјҡ 1 H' A2 q0 H( g2 u" ^ ]! }
04 жө·жҙӢеҫ®з”ҹзү©е®Ҹеҹәеӣ з»„ж•°жҚ®еә“
2 H* ^9 r6 Z: L
: U# a- o }6 F) ]- Z* | з»ҸеҺҶ35дәҝе№ҙзҡ„иҝӣеҢ–пјҢжө·жҙӢеҫ®з”ҹзү©еңЁжө·жҙӢз”ҹзү©зҡ„еӨҡж ·жҖ§гҖҒдё°еәҰе’ҢеҠҹиғҪд»Ји°ўдёӯеҚ жҚ®дәҶжңҖдё»иҰҒйғЁеҲҶпјҢ并еңЁжүҖжңүе…¶д»–жө·жҙӢз”ҹзү©з”ҹеӯҳе’ҢеҸ‘еұ•дёӯеҸ‘жҢҘзқҖеҹәзЎҖзҡ„дҪңз”ЁпјҢд№ҹжҳҜжө·жҙӢз”ҹжҖҒзҺҜеўғзі»з»ҹеӨҡж ·жҖ§е’ҢеҒҘеә·зӣ‘жөӢзҡ„йҮҚиҰҒз”ҹзү©ж Үеҝ—зү©гҖӮжҲ‘们ж•ҙеҗҲдәҶNCBIгҖҒEBIгҖҒJGIзӯүж•°жҚ®еә“дёӯзҡ„жө·жҙӢеҫ®з”ҹзү©е®Ҹеҹәеӣ з»„ж•°жҚ®пјҢиҺ·еҫ—дәҶ30,000еӨҡдёӘж ·жң¬пјҢиҰҶзӣ–3,100еӨҡдёӘйҮҮж ·зӮ№зҡ„иҝ‘200Tbж•°жҚ®пјҢе…¶дёӯеӨ§йғЁеҲҶж ·жң¬дҪҚдәҺеҶ…йҷҶжҲ–иҝ‘жө·ең°еҢәпјҢиҝңжҙӢе’Ңж·ұжө·ж ·е“Ғзҡ„ж•°жҚ®иҫғе°‘пјҲеӣҫ10пјүгҖӮжҲ‘们еҜ№иҝҷдәӣж ·жң¬зҡ„е®Ҹеҹәеӣ з»„ж•°жҚ®йҮҚж–°иҝӣиЎҢдәҶз»„иЈ…пјҢжһ„е»әдәҶиҝ„д»ҠдёәжӯўжңҖе®Ңж•ҙзҡ„жө·жҙӢеҫ®з”ҹзү©еҹәеӣ з»„ж•°жҚ®еә“пјҢеҫ—дәҶеҲҶеёғеңЁ125дёӘй—Ёзҡ„и¶…иҝҮ20,000дёӘй«ҳиҙЁйҮҸзҡ„жө·жҙӢз»ҶиҸҢеҹәеӣ з»„ (Metagenome-Assembled Genomes, MAGs)гҖӮе…¶дёӯпјҢзәҰ69%жҳҜжңӘеҲҶзұ»зҡ„жҪңеңЁж–°з§Қеҹәеӣ з»„пјҢеҢ…жӢ¬13дёӘеҖҷйҖүж–°й—Ёеҹәеӣ з»„гҖӮжҲ‘们иҝҳиҺ·еҫ—дәҶи¶…иҝҮ 2000 дёӘеҸӨз»ҶиҸҢеҹәеӣ з»„пјҢе…¶дёӯзәҰ 74% жҳҜжңӘеҲҶзұ»зҡ„еҹәеӣ з»„пјҢеҢ…жӢ¬ 2 дёӘеҖҷйҖүж–°й—Ёеҹәеӣ з»„гҖӮеҹәдәҺжө·жҙӢеҫ®з”ҹзү©еҹәеӣ з»„ж•°жҚ®еә“пјҢжҲ‘们иҝӣдёҖйғЁеҲҶеҜ№иө·еҹәеӣ з»„зү№еҫҒиҝӣиЎҢдәҶжҜ”иҫғпјҢе…¶дёӯзІҳз»ҶиҸҢпјҲMyxococcotaпјүе№іеқҮеҹәеӣ з»„жңҖеӨ§пјҢGCеҗ«йҮҸжңҖй«ҳпјӣиҖҢеҹәеӣ з»„жңҖе°Ҹзҡ„жө·жҙӢеҫ®з”ҹзү©дёәPatescibacteriaпјҢд»…жңү0.80MbпјҢжҳҜзІҳз»ҶиҸҢзҡ„1/5гҖӮжӯӨеӨ–жҲ‘еҸ‘зҺ°жө·жҙӢз»ҶиҸҢзҡ„GC еҗ«йҮҸдёҺеҹәеӣ з»„еӨ§е°Ҹд№Ӣй—ҙеӯҳеңЁжҳҫи‘—зҡ„дёүи§’еҪўе…ізі»пјҢеңЁОұ-еҸҳеҪўиҸҢе’Ңж”ҫзәҝиҸҢдёӯе°Өе…¶жҳҺжҳҫгҖӮжҲ‘们еңЁиҝҷдёӨз§Қеҫ®з”ҹзү©дёӯеҸ‘зҺ°пјҢеҪ“еҹәеӣ з»„еӨ§е°Ҹиҫғе°Ҹж—¶пјҢGCеҗ«йҮҸйҡҸзқҖеҹәеӣ з»„еӨ§е°Ҹзҡ„еўһеҠ иҖҢжҳҫи‘—еўһеҠ пјҢдҪҶдёҖж—ҰGCеҗ«йҮҸжҺҘиҝ‘70%еҗҺпјҢйҡҸзқҖеҹәеӣ з»„еӨ§е°ҸGCеҗ«йҮҸзҡ„дёҚеҸ—еҪұе“ҚпјҢжҸӯзӨәдәҶжө·жҙӢеҫ®з”ҹзү©GCеҗ«йҮҸзҡ„еҸҳеҢ–规еҫӢпјҲеӣҫ11пјүгҖӮжһ„е»әжӢҘжңүжө·жҙӢеҫ®з”ҹзү©еҹәеӣ з»„ж•°жҚ®еә“пјҢдёәиҝӣдёҖжӯҘзҡ„жө·жҙӢеҫ®з”ҹзү©еӨҡж ·жҖ§е’ҢеҠҹиғҪеӨҡж ·жҖ§з ”究гҖҒжҙ»жҖ§зү©иҙЁжҢ–жҺҳе’Ңдә§дёҡеҢ–еә”з”ЁзӯүжҸҗдҫӣдәҶйҮҚиҰҒзҡ„иө„жәҗгҖӮ + z' i- K: L) ]4 H5 K0 c
еӣҫ10. жө·жҙӢеҫ®з”ҹзү©е®Ҹеҹәеӣ з»„ж ·жң¬йҮҮж ·еҲҶеёғеӣҫ11. жө·жҙӢеҫ®з”ҹзү©зҡ„еҹәеӣ з»„еӨ§е°Ҹе’ҢGCеҗ«йҮҸзү№еҫҒжҜ”иҫғв–¶ йў„еҚ°ж–Үз« дҝЎжҒҜпјҡ 7 }2 q1 A: X' k7 ~; I; H( i
https://biorxiv.org/content/10.1101/2021.10.26.465843v1 5 `8 ?( m# P e2 c
в–¶ зӣёе…іж•°жҚ®йӣҶдҝЎжҒҜпјҡ + K$ s8 Y/ d z4 T
д»Һз”ҹе‘Ҫд№Ӣж ‘дёӯеҸҜд»ҘзңӢеҲ°зү©з§Қд»ҺеҚ•з»ҶиғһеҲ°еӨҡз»ҶиғһгҖҒд»Һз®ҖеҚ•еҲ°еӨҚжқӮзҡ„зҘһеҘҮиҝӣеҢ–пјҲеӣҫ12пјүгҖӮзҺ°еңЁе·Іжңүй’ҲеҜ№дёҚеҗҢзҫӨдҪ“зҡ„еҮ дёӘеӨ§еһӢеҹәеӣ з»„и®ЎеҲ’пјҢдҫӢеҰӮ VGPгҖҒB10KгҖҒFish10Kе’Ң10KPпјҢдҪҶзјәд№Ҹе…ідәҺжө·жҙӢеҫ®з”ҹзү©е’ҢиҪҜдҪ“еҠЁзү©зҡ„еӨ§еһӢеҹәеӣ з»„и®ЎеҲ’гҖӮдёәжӯӨпјҢйқ’еІӣеҚҺеӨ§еҹәеӣ з ”з©¶йҷўдёҺдёӯеӨ–жңәжһ„е…ұеҗҢжҺЁеҮәдәҶM10K+е’ҢGOMPдёӨеӨ§йЎ№зӣ®д»ҘеЎ«иЎҘиҝҷдәӣз©әзҷҪгҖӮе…¶зӣ®ж ҮжҳҜеңЁеҚҒе№ҙеҶ…еҜ№10,000з§ҚиҪҜдҪ“еҠЁзү©е’Ң100,000дёӘжө·жҙӢе®Ҹеҹәеӣ з»„ж ·жң¬иҝӣиЎҢжөӢеәҸгҖӮжңӘжқҘпјҢйҡҸзқҖйЎ№зӣ®зҡ„жҺЁиҝӣпјҢи¶ҠжқҘи¶ҠеӨҡзҡ„зү©з§Қе°Ҷиў«еӯҳеӮЁеңЁвҖңж•°еӯ—дҝЎжҒҜвҖқдёӯгҖӮиҝҷдәӣж•°еӯ—з”ҹе‘ҪйЎ№зӣ®зҡ„жңҖз»Ҳе®ҢжҲҗпјҢе°ҶеӨ§еӨ§еҠ еҝ«з”ҹе‘Ҫ科еӯҰзҡ„иҝӣжӯҘпјҢжҺЁеҠЁз”ҹзү©дә§дёҡзҡ„еҝ«йҖҹеҸ‘еұ•гҖӮйқ’еІӣеҚҺеӨ§еҹәеӣ з ”з©¶йҷўж¬ўиҝҺжӣҙеӨҡеҜ№иҝҷдәӣж•°жҚ®еә“ж„ҹе…ҙи¶Јзҡ„з ”з©¶дәәе‘ҳеҠ е…ҘдёҖиө·жҢ–жҺҳиҝҷдәӣж•°жҚ®гҖӮ
$ l2 L5 P- E9 c6 \& t) s еӣҫ12. з”ҹе‘Ҫд№Ӣж ‘дҝЎжҒҜжқҘжәҗпјҡвҖңйқ’еІӣеҚҺеӨ§вҖқе…¬дј—еҸ·
j7 Z7 R8 C& g9 G2 X4 _
7 y6 b# x) f' j8 m
, w) ?# \! E1 W) G" x
; |$ [4 n- J+ c* a% n0 B, R# \' t: @, A
| 

