|
: O1 [" |4 B7 b$ K! {% c& Q$ D/ C
原标题:自然资源部海洋三所邵宗泽团队在Anal Chem发文报道海洋微生物活性物质挖掘的“代谢组学-分子网络”综合策略
9 r) Q @7 [0 h/ c0 Y' V* r0 W7 } 海洋微生物是活性物质的重要来源。但微生物代谢物组成复杂,活性化合物往往丰度很低,无目标的分离纯化过程繁琐、低效。 9 ?2 T( J$ ~1 _# I! C W% ~ P4 }+ G! W
近日,自然资源部第三海洋研究所邵宗泽研究员团队,立足海洋微生物菌种保藏管理中心(MCCC)丰富的芽孢杆菌资源,从中挖掘对高致死性弧菌(vp-HL,可引起对虾“玻璃苗病”)有抑制活性的物质。通过将代谢组学、分子网络以及质谱结构相似性网络注释平台相结合,建立了一种海洋微生物活性物质高效挖掘的“代谢组学-分子网络”综合策略,实现了活性物质寻找过程从“大海捞针”式向“顺藤摸瓜”式的转变。相关研究成果发表在分析化学领域顶级学术期刊美国化学会旗下《Analytical Chemistry》(中科院一区TOP期刊)。 " p; \2 h' _2 _- |: L
研究人员对MCCC库藏海洋芽孢杆菌进行了广泛的活性筛选,从中发现了高活性菌株。以抗菌活性作为表型,运用代谢组学工具发现了造成菌株高活性的“生物标志物”分子。运用分子网络工具,预测活性物质是一类amicoumacin分子。进一步,在分子网络工具指导下,研究人员针对性获取了amicoumacin分子,并利用高分辨质谱、一维和二维核磁共振谱解析了其分子结构,验证了其抑制vp-HL的活性。分离结果证实了代谢组学与分子网络的分析预测,形成了闭环。 - `, M6 q' _( H$ O3 E( T& i# T
这项工作揭示了海洋芽孢杆菌来源代谢物对抗新型病原菌的潜力。更重要的,这一研究提出了一种综合策略,利用代谢组学和分子网络的优势,不经系统性的分离即可高效预测活性化合物类型,从而揭示粗提物中的“暗物质”。这一策略通过精准定位分离过程中的目标,克服了分离过程的随机性,提高了天然产物发现的效率和经济性,将在海洋微生物资源应用潜力的高效挖掘与评价中获得重要应用。
" Y( l, _7 E6 Y% ]% V 海洋三所夏金梅副研究员及其硕士生似鸿坤为论文共同第一作者,海洋三所王伟毅研究员和邵宗泽研究员为论文共同通讯作者。该工作得到了国家重点研发计划、国家自然科学基金面上项目、所基本科研业务费和厦门南方海洋研究中心项目等的资助。
3 P7 y# M% S+ @5 s& ~ 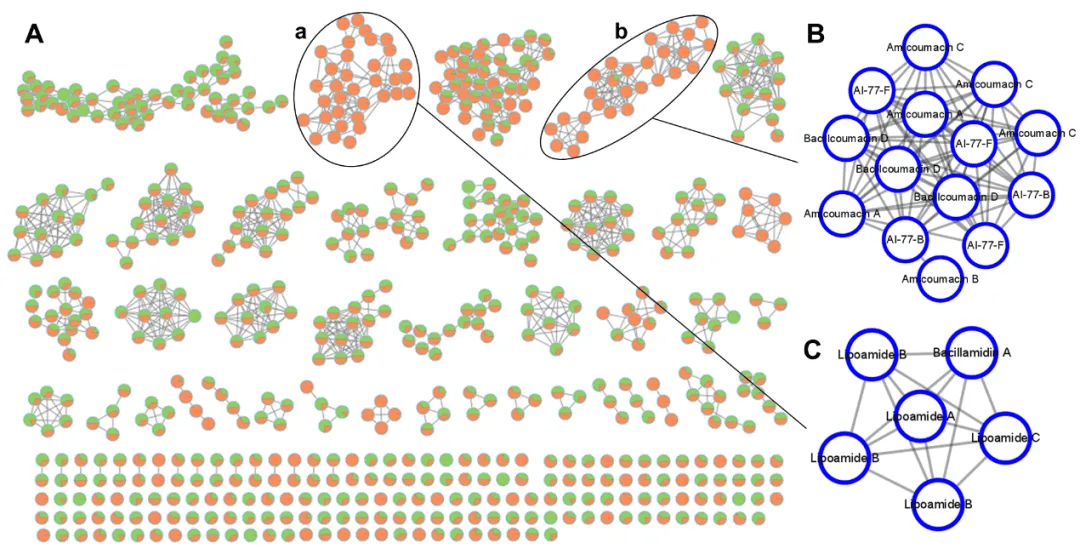
p& H3 u* N! B3 o
图1 分子网络预测和指认仅在高活性菌株中产生的化合物
+ P9 ]* ~+ ?0 A' p5 ^ c 点击左下“阅读原文”访问全文链接返回搜狐,查看更多
' ?7 y s2 ~+ V j1 R+ O* S+ F' L
9 ^0 n- d1 b2 }5 p 责任编辑: 4 l. Q; z j8 w* R- F
+ V- d0 m* K X5 r" p p) ]
% }, \% a4 e9 @' T/ ` |